
引物设计软件oligo 6破解版 win7 64位
分享到:
oligo6破解版是一款强大的引物设计软件,能够让我们的生物行业的用户轻松的进行各种引物的分析,在引物设计和分析可以说是非常著名且好用的。软件根据nearest neighbor thermodynamic values方法计算寡核苷酸的杂交温度和二级结构,不但如此,oligo6破解版还通过从一个序列中搜索、选择寡核苷酸而广泛应用于PCR、DNA测序、定向诱变及各种杂交中,是众多分子生物学行业人士必备的软件。
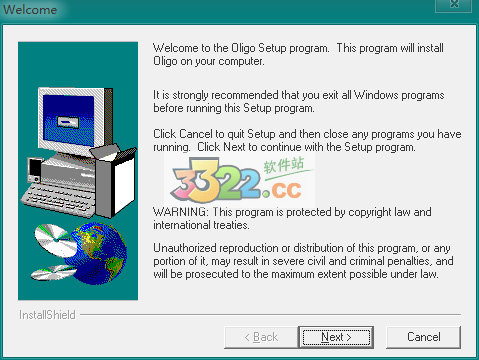
2、然后运行Setup.exe进行安装。
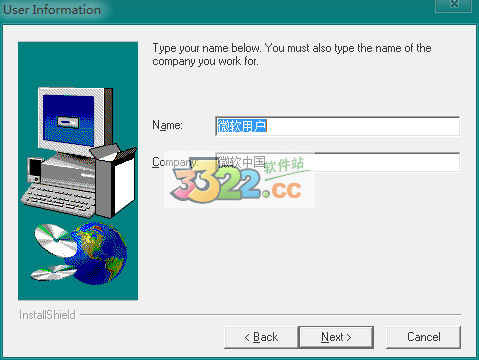
3、输入姓名和公司名,也可以不填,直接默认设置。
4、选择安装路径,用户根据自身选择安装路径。
5、点击next开始安装。
6、完成安装开始破解。
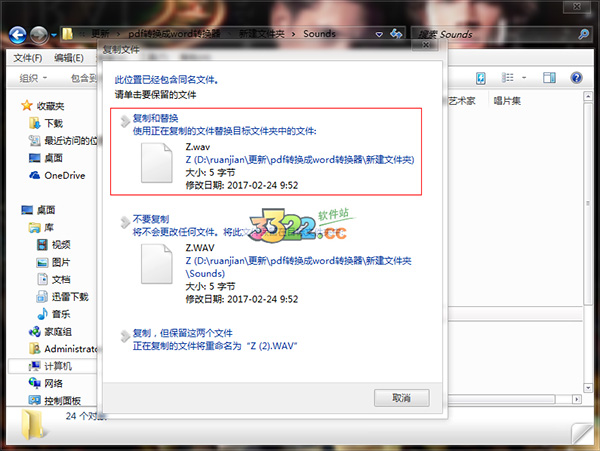
2、再将小编准备的破解补丁“Z.wav”复制到sound文件夹替换原文件即可完成破解。
2、在打开的窗口总,选择FreqSeq文件夹打开。
3、选择drosfr或者其他一个文件打开。
4、出现以下窗口,点击window-Tile。
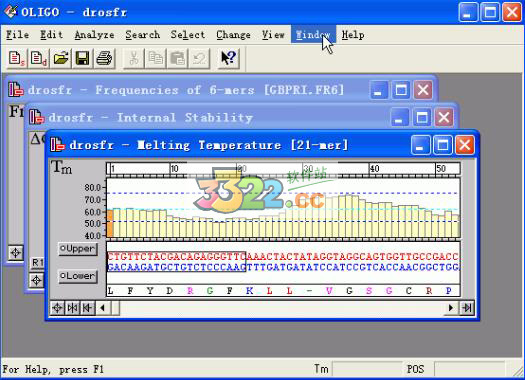
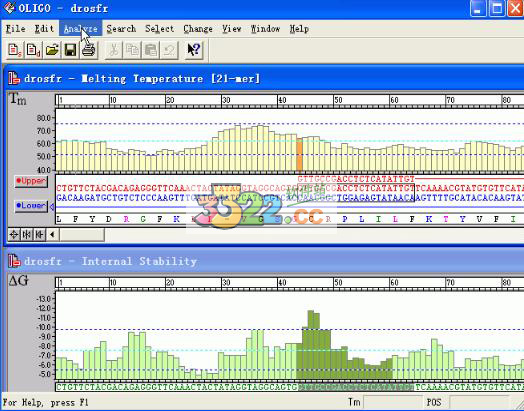
5、启动界面显示的三个指标分别为Tm、ΔG 和Frq,其中Frq 是6.22 版本的新功能,为邻近6至7 个碱基组成的亚单位在一个指定数据库文件中的出现频率。该频率高则可增加错误引发的可能性。因为分析要涉及多个指标,起动窗口的cascade 排列方式不太方便,可从windows菜单改为tili 方式。如果觉得太拥挤,可去掉一个指标,如Frq,这样界面的结构同于Oligo5.0,只是显示更清楚了。
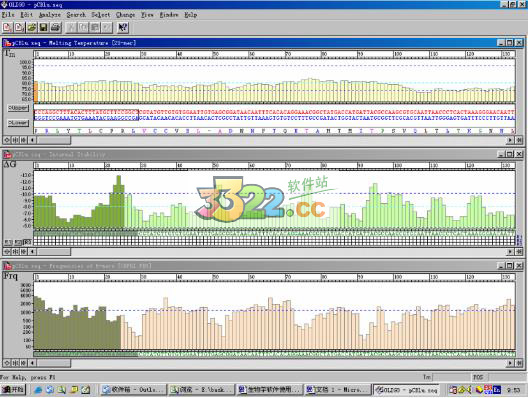
在设计时,可依据图上三种指标的信息选取序列,如果觉得合适,可点击Tm 图块上左下角的Upper 按钮,选好上游引物,此时该按钮变成,表示上游引物已选取好。下游引物的选取步骤基本同上,只是按钮变成Lower。G 值反映了序列与模板的结合强度,最好引物的G 值在5’端和中间值比较高,而在3’端相对低。
Tm 值曲线以选取72℃附近为佳,5’到3’的下降形状也有利于引物引发聚合反应。Frq 曲线为“Oligo 6”新引进的一个指标,揭示了序列片段存在的重复机率大小。选取引物时,宜选用3’端Frq 值相对较低的片段。
6、再点击Search,再点“for primers and probes”或使用快捷键F3.
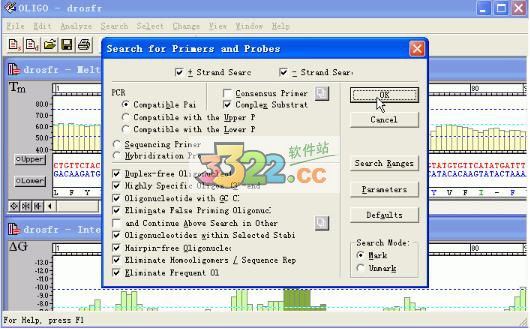
7、出现以下窗口点击OK就完成了,当然你也可以进行一些修改。
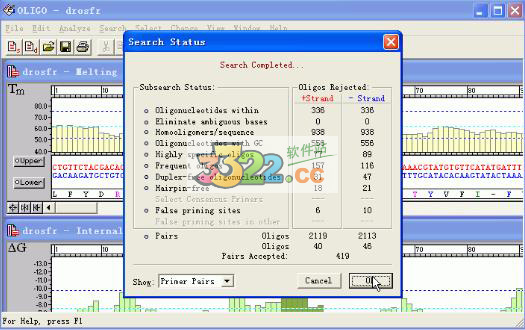
8、出现Search Status窗口,点击OK。
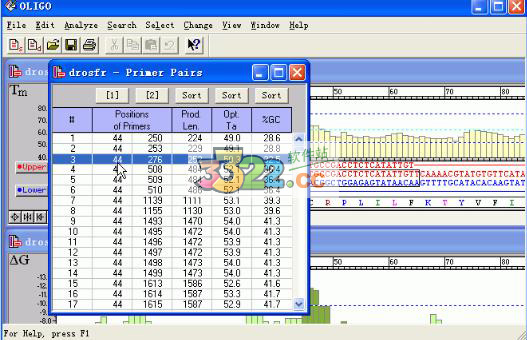
9、出现Primer pairs窗口,#代表引物对的编号,以此为引物对所处的位置、产物的长度、最适合的退火温度、和GC的百分比含量。
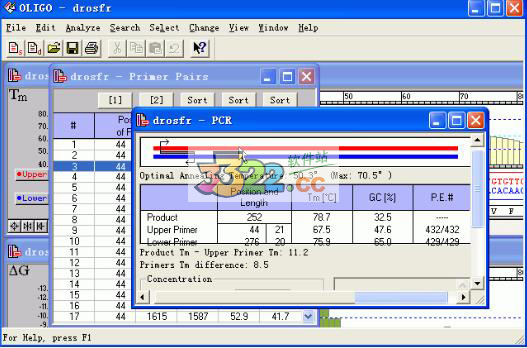
10、点击任何一行出现“PCR”窗口,告知你扩增片段的位置,最适合的退火温度等等信息。
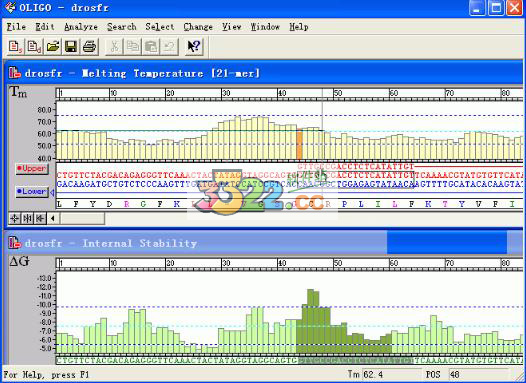
11、关掉“PCR”窗口和“Primer Pairs”窗口回到原来的窗口你就能够看到你引物的序列和位置,图中鼠标所指即为引物序列。
12、至此引物设计已经完成,可以用“Analyse”菜单进行分析,比如有无引物二聚体、发卡结构等等。
当上下游引物全选好以后,需要对引物进行评价并根据评价对引物进行修改。首先检查引物二聚体尤其是3’端二聚体形成的可能性。需要注意的是,引物二聚体有可能是上游或下游引物自身形成,也有可能是在上下游引物之间形成(cross dimer)。二聚体形成的能值越高,越不符合要求。一般的检测(非克隆)性PCR,对引物位置、产物大小要求较低,因而应尽可能选取不形成二聚体或其能值较低的引物。第二项检查是发夹结构(hairpin);与二聚体相同,发夹结构的能值越低越好。一般来说,这两项结构的能值以不超过4.5 为好。
当然,在设计克隆目的的PCR 引物时,引物两端一般都添加酶切位点,必然存在发夹结构,而且能值不会太低。这种PCR 需要通过灵活调控退火温度以达到最好效果,对引物的发夹结构的检测就不应要求太高。第三项检查为GC 含量,以45-55%为宜。有一些模板本身的GC 含量偏低或偏高,导致引物的GC 含量不能被控制在上述范围内,这时应尽量使上下游引物的GC 含量以及Tm 值保持接近,以有利于退火温度的选择。

oligo6破解版安装教程
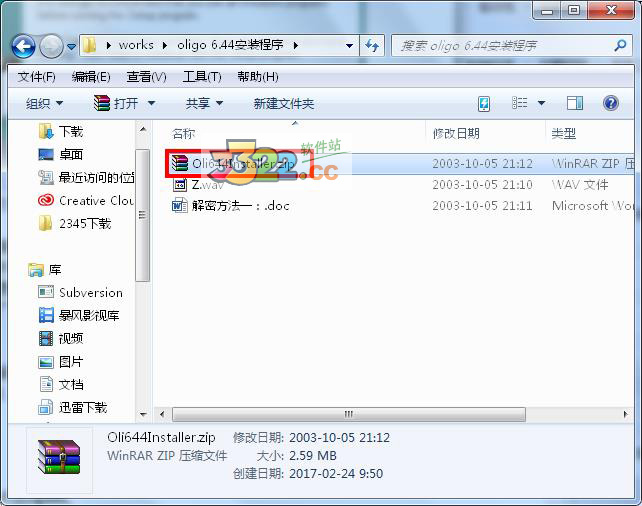
1、首先解压缩安装包,在解压其中的“Oli644Installer.zip”,这个压缩包是安装程序。
2、然后运行Setup.exe进行安装。

3、输入姓名和公司名,也可以不填,直接默认设置。

4、选择安装路径,用户根据自身选择安装路径。

5、点击next开始安装。

6、完成安装开始破解。

oligo6破解教程
1、首先运行软件,出现注册窗口,将其关闭。
2、再将小编准备的破解补丁“Z.wav”复制到sound文件夹替换原文件即可完成破解。

oligo6破解版使用方法
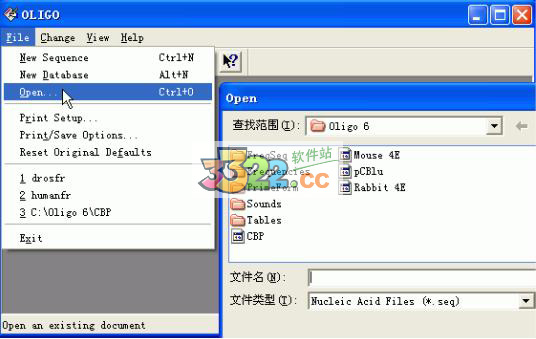
1、单击file菜单,再点击open。
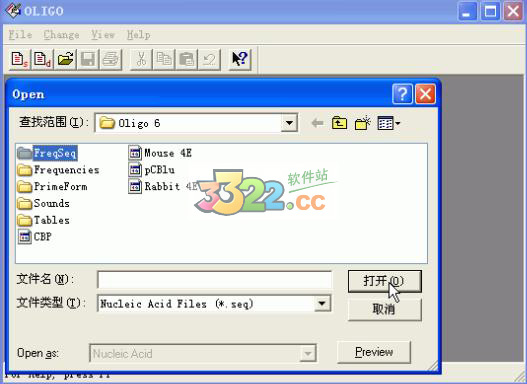
2、在打开的窗口总,选择FreqSeq文件夹打开。

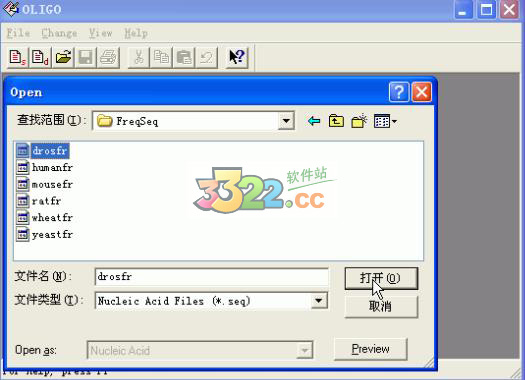
3、选择drosfr或者其他一个文件打开。

4、出现以下窗口,点击window-Tile。

5、启动界面显示的三个指标分别为Tm、ΔG 和Frq,其中Frq 是6.22 版本的新功能,为邻近6至7 个碱基组成的亚单位在一个指定数据库文件中的出现频率。该频率高则可增加错误引发的可能性。因为分析要涉及多个指标,起动窗口的cascade 排列方式不太方便,可从windows菜单改为tili 方式。如果觉得太拥挤,可去掉一个指标,如Frq,这样界面的结构同于Oligo5.0,只是显示更清楚了。

在设计时,可依据图上三种指标的信息选取序列,如果觉得合适,可点击Tm 图块上左下角的Upper 按钮,选好上游引物,此时该按钮变成,表示上游引物已选取好。下游引物的选取步骤基本同上,只是按钮变成Lower。G 值反映了序列与模板的结合强度,最好引物的G 值在5’端和中间值比较高,而在3’端相对低。
Tm 值曲线以选取72℃附近为佳,5’到3’的下降形状也有利于引物引发聚合反应。Frq 曲线为“Oligo 6”新引进的一个指标,揭示了序列片段存在的重复机率大小。选取引物时,宜选用3’端Frq 值相对较低的片段。
6、再点击Search,再点“for primers and probes”或使用快捷键F3.

7、出现以下窗口点击OK就完成了,当然你也可以进行一些修改。

8、出现Search Status窗口,点击OK。

9、出现Primer pairs窗口,#代表引物对的编号,以此为引物对所处的位置、产物的长度、最适合的退火温度、和GC的百分比含量。

10、点击任何一行出现“PCR”窗口,告知你扩增片段的位置,最适合的退火温度等等信息。

11、关掉“PCR”窗口和“Primer Pairs”窗口回到原来的窗口你就能够看到你引物的序列和位置,图中鼠标所指即为引物序列。

12、至此引物设计已经完成,可以用“Analyse”菜单进行分析,比如有无引物二聚体、发卡结构等等。

当上下游引物全选好以后,需要对引物进行评价并根据评价对引物进行修改。首先检查引物二聚体尤其是3’端二聚体形成的可能性。需要注意的是,引物二聚体有可能是上游或下游引物自身形成,也有可能是在上下游引物之间形成(cross dimer)。二聚体形成的能值越高,越不符合要求。一般的检测(非克隆)性PCR,对引物位置、产物大小要求较低,因而应尽可能选取不形成二聚体或其能值较低的引物。第二项检查是发夹结构(hairpin);与二聚体相同,发夹结构的能值越低越好。一般来说,这两项结构的能值以不超过4.5 为好。
当然,在设计克隆目的的PCR 引物时,引物两端一般都添加酶切位点,必然存在发夹结构,而且能值不会太低。这种PCR 需要通过灵活调控退火温度以达到最好效果,对引物的发夹结构的检测就不应要求太高。第三项检查为GC 含量,以45-55%为宜。有一些模板本身的GC 含量偏低或偏高,导致引物的GC 含量不能被控制在上述范围内,这时应尽量使上下游引物的GC 含量以及Tm 值保持接近,以有利于退火温度的选择。
展开更多
引物设计软件oligo 6破解版 win7 64位下载地址
- 需先下载高速下载器:
- 专用下载:
- 其它下载:







































